python3-scipy-convolve2d图像函数卷积
发布时间:2021-08-22 22:28:08编辑:run阅读(7851)
使用scipy-convolve2d函数进行卷积.
理解卷积:
卷积是对两幅图像进行操作运算:一幅是输入图像,另一幅图像是作为输入图像的滤波器而产生输出图像的掩模(也称为核).
卷积滤波用于图像的空间频率特性的修正。它的工作原理是通过将所有相邻像素的加权重相加来确定中心像素的值,从而计算出输出图像像素的值。输出图像中的像素值是通过输入图像遍历核窗口来计算的。
为什么需要卷积图像?
卷积对输入图像使用了一种通用的滤波效果,这样做是为了在图像上使用合适的核实现各种效果(如平滑,锐化和浮雕),以及在边缘检测等操作中实现各种效果。
使用scipy信号模块的convolve2d函数进行卷积,scipy信号模块的convolve2d()函数可用于互相关。
对灰度图像应用卷积。
from skimage.io import imread
from skimage.color import rgb2gray
import matplotlib.pylab as pylab
import numpy as np
from scipy import signal
im = rgb2gray(imread(r'D:\image_processing\image_two\1dd.jpg'))
print(np.max(im))
print(im.shape)
# 指定默认字体
pylab.rcParams['font.sans-serif'] = ['KaiTi']
# 解决保存图像是负号'-'显示为方块的问题
pylab.rcParams['axes.unicode_minus'] = False
blur_box_kernel = np.ones((3, 3)) / 9
edge_laplace_kernel = np.array([[0,1,0],[1,-4,1],[0,1,0]])
im_blurred = signal.convolve2d(im, blur_box_kernel)
im_edges = np.clip(signal.convolve2d(im, edge_laplace_kernel), 0, 1)
fig, axes = pylab.subplots(ncols=3, sharex=True, sharey=True, figsize=(18, 6))
axes[0].imshow(im, cmap=pylab.cm.gray)
axes[0].set_title('原始图像', size=20)
axes[1].imshow(im_blurred, cmap=pylab.cm.gray)
axes[1].set_title('方框模糊', size=20)
axes[2].imshow(im_edges, cmap=pylab.cm.gray)
axes[2].set_title('拉普拉斯边缘检测', size=20)
for ax in axes:
ax.axis('off')
pylab.show()
卷积模式, 填充值和边界条件。
卷积模式,填充值和边界条件取决于对边缘像素做怎么样的处理,这里有3个参数:mode,boundary和fillvalue,它们都将传递给scipy的convolve2d()函数。
(1). mode='full'。默认模式,其中输出是输入的完全离散线性卷积。
(2). mode='valid'。忽略边缘像素,只计算所有相邻像素(不需要零填充的像素)。对于所有核(除了1 x 1),输出图像的大小都小于输入图像的大小。
(3). mode='same'。输出图像与输入图像大小相同,它以full输出为中心。
彩色图像的卷积。使用scipy.convolve2d()可以锐化彩色图像,这要求对每个图像通道分别进行卷积。使用emboss核和schar边缘检测复杂核对图像进行卷积。
from skimage.io import imread
import matplotlib.pylab as pylab
import numpy as np
from scipy import signal, misc, ndimage
# 指定默认字体
pylab.rcParams['font.sans-serif'] = ['KaiTi']
# 解决保存图像是负号'-'显示为方块的问题
pylab.rcParams['axes.unicode_minus'] = False
im = imread(r'D:\image_processing\image_two\qq1.jpg')
print(np.max(im))
sharpen_kernel = np.array([0, -1, 0, -1, 5, -1, 0, -1, 0]).reshape((3, 3, 1))
emboss_kernel = np.array(np.array([[-2, -1, 0], [-1, 1, 1], [0, 1, 2]])).reshape((3, 3, 1))
im_sharp = ndimage.convolve(im, sharpen_kernel, mode='nearest')
im_sharp = np.clip(im_sharp, 0, 255).astype(np.uint8)
im_emboss = ndimage.convolve(im, emboss_kernel, mode='nearest')
im_emboss = np.clip(im_emboss, 0, 255).astype(np.uint8)
pylab.figure(figsize=(10, 15))
pylab.subplot(311),pylab.imshow(im.astype(np.uint8)),pylab.axis('off')
pylab.title('原图', size=25)
pylab.subplot(312),pylab.imshow(im_sharp),pylab.axis('off')
pylab.title('卷积后的锐化图像', size=25)
pylab.subplot(313),pylab.imshow(im_emboss),pylab.axis('off')
pylab.title('卷积后的浮雕图像', size=25)
pylab.tight_layout()
pylab.show()
相关与卷积。与卷积运算非常相似,相关也接收一个输入图像和另一个核,计算像素邻域值与核值的加权组合,通过输入遍历核窗口,生成输出图像。唯一的区别是,与相关不同,卷积在计算加权组合之前会对核进行两次翻转(相对于水平轴和垂直轴)。
scipy信号模块的correlation2d()函数可用于相关。如果核是对称的,则相关类似于卷积;如果核不是对称的,为了获得与correlation2d()相同的结果,在将核置于图像中之前,必须将图像上下颠倒,左右翻转。
模版匹配与图像和模版之间的互相关。
使用与眼睛模块图像的互相关(使用带有图像的核进行互相关),可以找到图像中眼睛的位置。
import matplotlib.pylab as pylab
import numpy as np
from scipy import signal, misc, ndimage
# 指定默认字体
pylab.rcParams['font.sans-serif'] = ['KaiTi']
# 解决保存图像是负号'-'显示为方块的问题
pylab.rcParams['axes.unicode_minus'] = False
face_image = misc.face(gray=True) - misc.face(gray=True).mean()
template_image = np.copy(face_image)[300:365, 670:750]
template_image -= template_image.mean()
face_image = face_image + np.random.randn(*face_image.shape) * 50
correlation = signal.correlate2d(face_image, template_image, boundary='symm', mode='same')
y, x = np.unravel_index(np.argmax(correlation), correlation.shape)
fig, (ax_original, ax_template, ax_correlation) = pylab.subplots(3, 1, figsize=(6, 15))
ax_original.imshow(face_image, cmap='gray')
ax_original.set_title('原始图', size=25)
ax_original.set_axis_off()
ax_template.imshow(template_image, cmap='gray')
ax_template.set_title('眼睛模版', size=25)
ax_template.set_axis_off()
ax_correlation.imshow(correlation, cmap='afmhot')
ax_correlation.set_title('浣熊与模版的互相关', size=25)
ax_correlation.set_axis_off()
ax_original.plot(x, y, 'ro')
fig.show()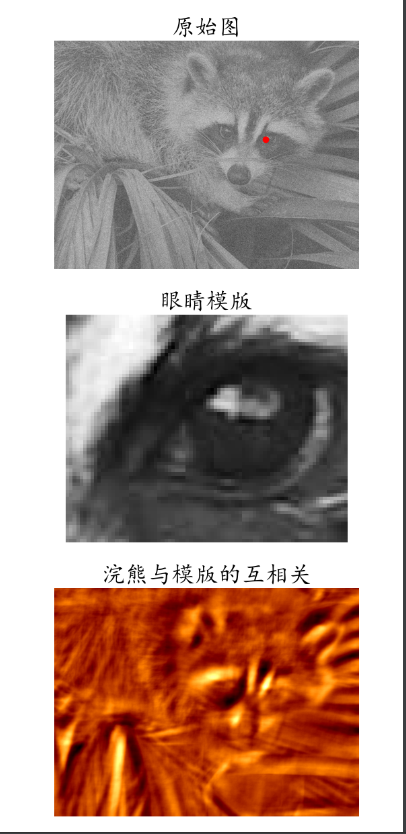
- openvpn linux客户端使用
52213
- H3C基本命令大全
52118
- openvpn windows客户端使用
42285
- H3C IRF原理及 配置
39151
- Python exit()函数
33655
- openvpn mac客户端使用
30613
- python全系列官方中文文档
29296
- python 获取网卡实时流量
24274
- 1.常用turtle功能函数
24160
- python 获取Linux和Windows硬件信息
22528
- LangChain 1.0-Agent中间件-实现闭环(批准-编辑-拒绝动作)
94°
- LangChain 1.0-Agent中间件-汇总消息
100°
- LangChain 1.0-Agent中间件-删除消息
108°
- LangChain 1.0-Agent中间件-消息压缩
100°
- LangChain 1.0-Agent中间件-多模型动态选择
164°
- LangChain1.0-Agent-部署/上线(开发人员必备)
321°
- LangChain1.0-Agent-Spider实战(爬虫函数替代API接口)
364°
- LangChain1.0-Agent(进阶)本地模型+Playwright实现网页自动化操作
359°
- LangChain1.0-Agent记忆管理
338°
- LangChain1.0-Agent接入自定义工具与React循环
378°
- 姓名:Run
- 职业:谜
- 邮箱:383697894@qq.com
- 定位:上海 · 松江
